目前,人类社会的可持续发展面临着众多的挑战和危机,涉及全球气候变暖、土壤与水体污染、抗生素耐药性以及生物多样性降低等方面。Science杂志在2021年将“我们可以阻止全球气候变化吗?”和“我们能把过量的二氧化碳存到何处?”等列入了全世界最前沿的125个科学问题。微生物、病毒和质粒等在地球元素循环中扮演着关键角色,其独特基因和代谢功能能够为提高人类的生产效率并改善生存条件提供帮助。深入认识微生物、病毒和质粒等的多样性、功能特征、环境影响,以及潜在的可利用资源,一直以来都是微生物生态学研究的核心内容。因此,阐明微生物与病毒等的互作机制和环境效应,对微生物多样性的形成和保护、新生物技术方法的开发和理解它们在气候变化中的角色和功能等都至关重要。
尽管学界在微生物资源利用方面已取得了一些进展,但要实现借助微生物来改善全球环境的愿景仍需跨越诸多障碍。这主要是因为我们对微生物、病毒和质粒等染色体外遗传物质的多样性和功能的了解尚不充分;对它们在生态系统中复杂的相互作用的理解和环境效应的认识仍然十分有限;以及当前的研究技术和方法仍然存在一定局限性等。
针对这些挑战,本团队聚焦土壤和淡水湿地等生境,应用宏基因组学、宏转录组学、DNA-SIP、实验室富集培养、基因编辑等先进技术,继续攻克上述科学障碍,扩展对微生物和病毒、质粒等染色体外遗传物质的多样性和功能的认识,揭示它们的互作机制和环境效应,挖掘可利用的微生物资源,为改善环境做出贡献(具体如下图所示)。
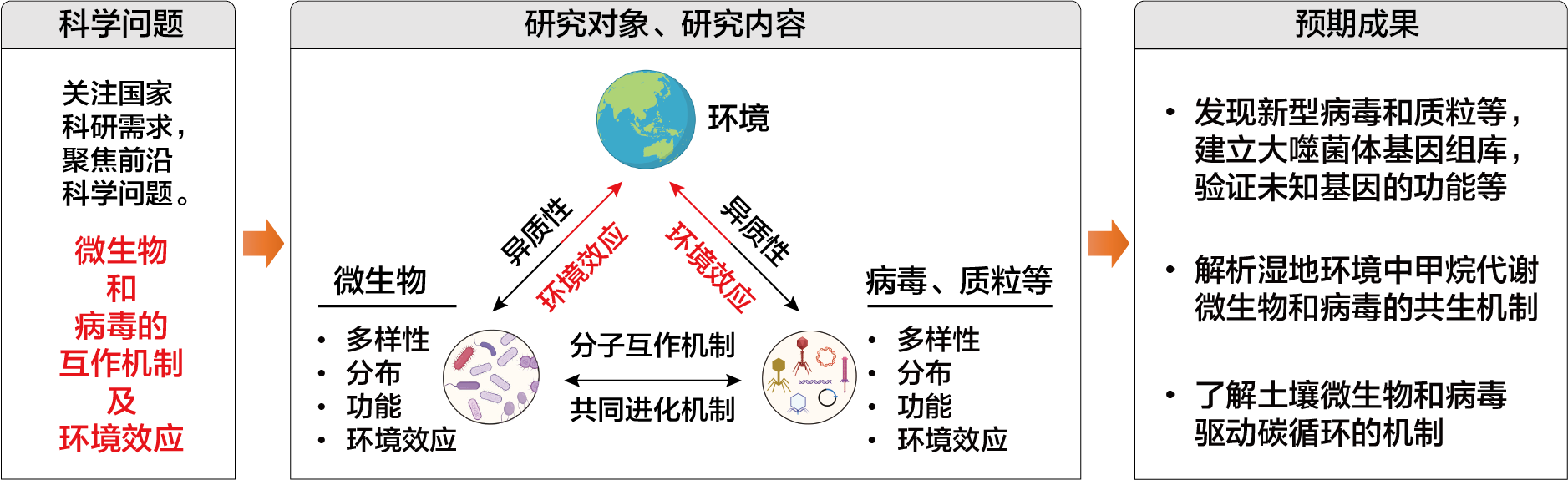
1. 染色体外遗传物质和可移动基因元件的鉴定及其多样性和功能研究。
在微生物领域,染色体外遗传物质(extrachromosomal genetic elements,ECEs)包括病毒、质粒以及其他尚未分类的遗传物质;而可移动基因元件(mobile genetic elements,MGEs)则涵盖了病毒、质粒、转座子以及其他未分类元件。近年来,科学家利用宏基因组发现了越来越多的 ECEs 和 MGEs,其中一些对于环境和人类的健康具有至关重要的作用。然而,学界对于它们的鉴定,特别是功能的研究,却一直存在着挑战。在过去5年的研究中,我们利用宏基因组学揭示了几类重要的 ECEs 和 MGEs 的分布和功能特征,例如 PmoC-phages 和 TnpBs-Fanzors(Nucleic. Acids. Res., 2023, gkad1053)。未来,本团队将继续利用多组学技术和实验验证等手段,探索更多未知且具有潜在重要意义的 ECEs 和 MGEs。具体来说,将围绕以下几个方面展开研究:
(1) 利用宏基因组学数据鉴定新型 ECEs 和 MGEs:宏基因组提供了大量不可分类的基因组数据,其中很多是潜在的新型 ECGs 和 MGEs。拟对公共数据库中某些特定生境的数据和实验室从头测序的数据进行挖掘,利用基因组比对、标记基因、机器学习等方法来鉴定未知的或者新型的 ECGs 和 MGEs,并对它们的多样性、分布、功能和生态位角色等进行系统分析,以揭示它们在地球元素循环和环境健康等方面的作用。
(2) 构建全球生境的大噬菌体基因组数据库:大噬菌体的生态和进化研究具有重要的意义,而目前已报道的大噬菌体基因组还很少(~2000个左右),这限制了对它们的认识。拟通过收集已报道的大噬菌体基因组,同时通过其开发的 COBRA 等工具从宏基因组数据中获取更多未报道的基因组,从而建立全球生境的大噬菌体基因组数据库。在此基础上,解释大噬菌体的多样性、分布和功能,并揭示它们的进化历史。
(3) 结合大数据分析和分子实验验证噬菌体的未知功能基因:噬菌体编码了很多未知功能的基因,它们通常与已知蛋白具有极低的序列相似性(甚至无相似性),但在三维结构上却可能存在相似之处。拟通过对大噬菌体编码的基因进行蛋白质家族聚类分析,然后选取一些未知的且为大部分基因组所编码的基因进行基因区域分析和 Alphafold 三维结构预测。基于结构预测推测出可能的蛋白功能,然后利用编码了该蛋白家族的可培养噬菌体进行基因敲除或沉默和噬菌斑分析等,来揭示该蛋白家族在噬菌体中的功能和意义。
2. 淡水湿地环境中微生物和病毒介导的物质能量循环与环境效应。
淡水湿地包括淡水湖泊、河流、沼泽、湿地草地、湿地滩涂等生境。尽管淡水湿地仅占地球陆地面积的不到8%,但它们却蕴含着丰富的微生物资源,在全球碳循环中具有重要作用,而且是甲烷排放的主要来源之一。了解淡水湿地中微生物、病毒和质粒等的多样性和功能等特征,将为我们监测和控制水污染以及甲烷的排放提供科学依据。基于此,计划开展以下几个方面的研究:
(1)淡水湖泊中的微生物、病毒和质粒等的动态特征及其在水华中的角色和功能:在淡水湖泊中,微生物、病毒和质粒等的群落动态变化对水华形成过程至关重要。我们将通过长期的和多深度的采样、细胞计数和高通量测序等技术手段,探索微生物、病毒和质粒等的在水体中的空间分布和季节变化规律等。利用宏基因组学和宏转录组学等方法,解析微生物、病毒和质粒等在水华中的功能作用、潜在的互作机制以及与藻类等其他生物体之间的相互联系等等。
(2)淡水湿地中甲烷代谢微生物和病毒等的多样性和活性:微生物和病毒等染色体外遗传物质(如 PmoC-phages,Borgs 等)是甲烷循环的关键调控因子。将针对不同类型的淡水湿地样品,利用宏基因组学、宏转录组学和 DNA-SIP 等方法,研究与甲烷代谢相关的微生物和病毒等的多样性和群落结构以及时空分布规律,深入探讨它们在甲烷的产生和消耗中的作用,从而理解其在淡水湿地甲烷循环中的贡献和调控机制。
(3)探索 PmoC-phages 的多样性、分布和基因组改造的可行性:PmoC-phages是甲烷氧化细菌噬菌体,已在湖泊、河流、土壤和污水处理厂等生境中被发现,因此可能在甲烷氧化细菌的生理功能和进化中发挥重要作用。将利用宏基因组系统调查不同环境中 PmoC-phages 的多样性、丰度和进化等,并基于实验室的富集或分离培养来解析其参与甲烷氧化的分子机制,了解其与甲烷氧化细菌的共生关系。同时尝试通过基因编辑技术,探索调控 PmoC-phages 与甲烷氧化细菌相互作用的可行性,以及其对甲烷循环的影响。
3. 介导土壤碳循环的微生物和病毒的多样性和机制研究。
在当前环境变化日益严峻的背景下,深入研究土壤微生物和病毒在碳循环中的作用至关重要。它们不仅是土壤生态系统中的重要组成部分,还直接影响着碳循环过程及其对气候变化的调节作用。了解其多样性、功能、互作关系、环境效应、及其应对环境变化的调节机制,有助于揭示土壤生态系统的稳定性和可持续性,为土壤资源的保护和管理提供科学依据。基于此,拟开展以下研究:
(1) 碳循环相关的微生物和病毒研究:利用宏基因组和宏转录组技术,深入挖掘土壤微生物和病毒的多样性和功能,特别是那些与碳循环密切相关的种类。通过室内实验并结合 DNA-SIP 等分析技术,追踪标记碳同位素以研究不同微生物和病毒群体在碳循环中的作用和相互关系。探索微生物和病毒在应对环境变化时的调节机制,以及它们对碳循环过程中关键基因的表达调控。
(2) RuBisCO基因的多样性,相应微生物的分离和固碳能力的评估:通过宏基因组和分子进化分析等技术,对土壤微生物中 RuBisCO 基因的多样性和进化历史进行深入分析。其次,采用微生物分离培养技术,从土壤样品中分离和鉴定不同 RuBisCO 基因型的微生物,并结合DNA-SIP 等技术评估其固碳能力等。对新型的 RuBisCO 基因进行蛋白表达,解析其蛋白结构和功能特征等。
(3) 探索通过基因编辑增强土壤微生物的固碳能力的可能性:基于上述两方面的研究结果,在实验室内利用 CRISPR-Cas 等技术,尝试对分离到的土壤微生物进行固碳相关基因的定向编辑,以提高其固碳能力。其次,在实验室条件下构建人工群落,通过宏基因组和宏转录组等技术,评估编辑后的微生物在群落中的生态功能和适应性等特征,并探索潜在的应用前景。
微生物是地球元素循环的核心驱动力,对生命和环境健康至关重要。了解微生物的生态和进化对揭示其在地球元素循环中的角色是不可或缺的。病毒作为地球上丰度最高的生物实体,对微生物群落具有深远影响,不仅能通过感染和裂解来控制宿主物种的数量,通过携带的辅助代谢因子增强宿主的代谢潜能,还可以促进新物种形成和增加遗传多样性,是微生物适应环境和生物进化的关键因素之一。 因此,在研究微生物驱动元素循环时,环境中的病毒也是不可忽视的。我们以宏基因组学、宏转录组学和生物信息学为主要研究手段,聚焦微生物和病毒在驱动地球元素循环中的作用,开展了关于其物种多样性、分布、功能、基因活性和进化历史等的研究工作,主要成果包括:(1)以矿业废弃物相关生境为研究对象,揭示了其中微生物的物种多样性、功能和活性等特征,为环境污染的监控和治理提供了科学依据;(2)对多个特殊的噬菌体(即细菌病毒)类群的多样性、分布、功能、活性和进化历史进行了报道,拓展了学界对噬菌体的认识;其中,首次报道了噬菌体直接参与甲烷氧化过程,为控制甲烷排放提供了新的研究思路;(3)针对宏基因组学的自身缺陷,开发了获取高质量基因组的方法和工具。
1. 微生物的物种多样性、功能和活性研究:以矿业废弃物相关生境为例
矿产资源是社会生产发展的重要物质基础,但伴随采矿活动而产生的各种废弃物,给生态环境造成了显著压力。在自然条件下,尾矿等矿业废弃物通过微生物介导的金属硫化物的氧化可产生低 pH 和高浓度重金属的水体,即酸性矿山废水(acid mine drainage, AMD)。
针对矿业废弃物的自然酸化过程,对我国南方有色金属矿业废弃物生境中的微生物介导 AMD 产生的过程进行了系统性的研究。首先,揭示了尾矿从酸化起始到 AMD 产生这一过程中的微生物群落结构和功能的变化特征,明确了特定微生物类群在尾矿酸化早期驱动碳氮固定和硫氧化的关键作用(Environ. Microbiol. 2013, 15, 2431-2444),为针对矿业废弃物的前期治理提供了科学依据。同时,通过比较不同矿区的微生物群落(ISME J. 2015, 9, 1579-1592),以及对单个群落进行深度分析(ISME J. 2015, 9, 1280-1294),揭示了AMD环境中优势物种和稀有物种在转录水平上对环境变化的响应机制。基于上述研究,总结了 AMD 微生物驱动元素循环的模式,提出了利用 AMD 土著微生物来进行污水处理的理论模型(Curr. Opin. Biotechnol. 2016, 38, 150-158)。此外,揭示了AMD中的重要古菌类群 Micrarchaeota 和 Parvarchaeota在多种生境中的广泛分布,代谢多样性及其进化历史,并在对其基因组特征进行充分了解的前提下,设定特定条件在实验室内首次成功地对它们进行了富集培养(ISME J. 2018, 12, 756-775,)。
为了抑制或减缓 AMD 的产生,通常利用人工湖将矿业废弃物进行覆水封存。为了了解其中的微生物学过程,对加拿大某矿区的人工湖的相关数据进行了分析。此项目进行了多年连续的水体采样(2015-2018年),并对其中的多种硫化物进行了测定,结合宏基因组和宏转录组分析,以了解其中的微生物在碳、氮、硫等元素循环过程的角色和活性。分析结果显示,在有氧条件下,一个新型的 Halothiobacillus 类群通过完整的 Sox 通路来进行硫化物的氧化,从而产生低 pH 的水体;而在低氧或无氧的条件下,这一过程则是由 Thiobacillus 类群通过非完整的 Sox 通路和 rDSR 途径通过耦合硝酸盐还原来驱动的。这一结果揭示了在近中性条件下,硫化物氧化和酸度生成的模式,在防止可能影响下游环境的硫化物排放方面具有重要的应用意义。

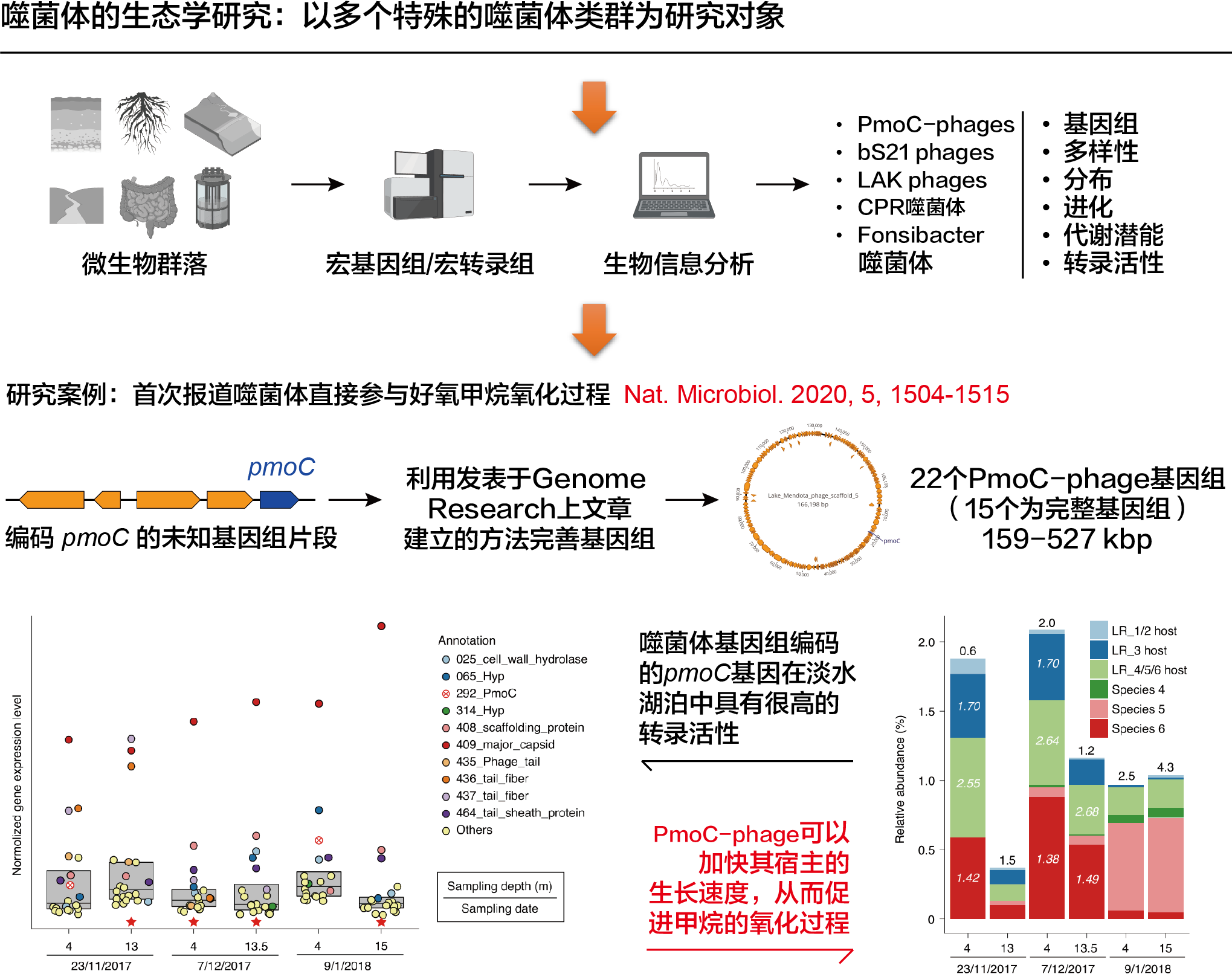
2. 噬菌体的物种多样性、分布、功能、活性和进化历史研究
噬菌体(即细菌病毒)不仅在宿主微生物的进化等方面发挥着重要作用,同时其携带的辅助代谢因子往往可以行使某些重要的代谢功能,如光合作用、氨氧化等等。然而,由于噬菌体具有极大的多样性,目前对它们这方面的了解还只是“冰山一角”。
甲烷氧化细菌通过甲烷单加氧酶来进行甲烷的氧化,从而减少排入大气中的甲烷数量,因此对它们的探索有助于了解大气变暖等问题。甲烷单加氧酶包括 PmoA,PmoB 和 PmoC 三个亚基,有证据表明 PmoC 是其中最重要的亚基,然而该基因此前从未在噬菌体中被发现。发现来自油砂尾矿处理人工湖的宏基因组数据中,有一些基因组片段编码了 PmoC 亚基。然而这些基因片段长度通常都比较短,而且其分类信息是未知的,因此利用其开发的提高基因组质量的方法,通过得到完整的基因组来获得更多的信息。对数十个宏基因组进行分析,恢复了22个编码 PmoC 亚基的基因组,其中15个是完整的基因组(大小为159-527 kbp),标记分子分析表明它们都是噬菌体基因组(命名为PmoC-phages)。基于 CRISPR-Cas 和噬菌体-宿主共存模式分析,明确了 PmoC-phages 的宿主是好氧甲烷氧化菌。噬菌体编码的 pmoC 基因与其宿主的该基因具有很高的相似度,表明该基因很可能是从其宿主获得的。宏转录组数据分析发现,噬菌体 pmoC 基因往往具有很高的转录活性,同时可以加快宿主的生长速度,从而促进甲烷的氧化过程。这是首次发现噬菌体可以直接参与甲烷氧化过程,这一工作以第一作者身份发表于 Nature Microbiology (2020)。
此外,报道了 Fonsibacter(陆地环境中丰度最高的微生物属)的首个噬菌体基因组,并揭示了它们从海洋到陆地环境的进化历史 (mSystems, 2019) 。还发现某些小型细菌(即CPR)利用多种类型的 CRISPR-Cas 系统来抵抗病毒(Front. microbiol. 2019, 10, 928);探索了大噬菌体编码的核糖体蛋白 S21 在噬菌体复制后期的作用(ISME Commun. 2022, 2, 31);揭示了 LAK 大噬菌体在动物肠道中的分布,多样性和进化特征等(iScience 2021, 24, 102875)。另外,以第三作者身份参与揭示了大噬菌体的广泛分布和特殊的“生命”机制(Nature. 2020, 578, 425-431)。 综上,通过对多个特殊的噬菌体类群进行了生态学研究,揭示了它们的基因组大小、多样性、分布、功能和进化历史等,拓展了学界对噬菌体的认识,为甲烷排放控制提供了新的研究思路。
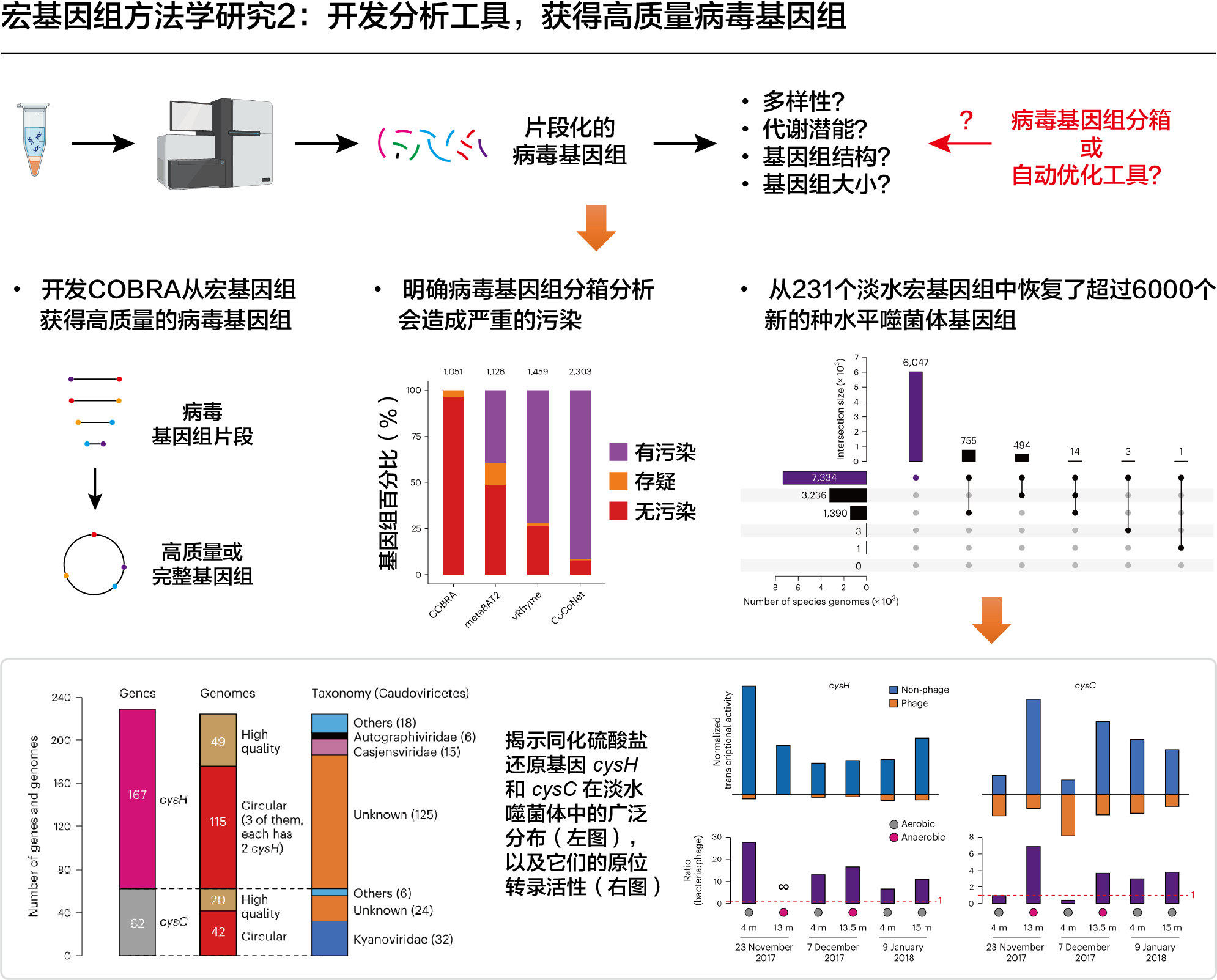
3. 优化宏基因组学数据的方法和工具开发
高质量的基因组是微生物和病毒生态学研究的基础,对确定它们在生态系统中的功能和生态位角色是不可或缺的。由于大部分的微生物和病毒不能被分离培养和测序,这限制了学界对它们的了解。近20年来,不依赖于培养的宏基因组学成为了重要的研究手段之一。宏基因组学通过从样品中提取总DNA、测序和拼接等来获得基因组(即metagenome-assembled genomes, MAGs),并利用生物信息学分析来揭示微生物和病毒的多样性、功能和生态学角色等特征。然而,宏基因组获得的 MAGs 具有一些缺陷,这限制了宏基因组学的进一步发展和应用。
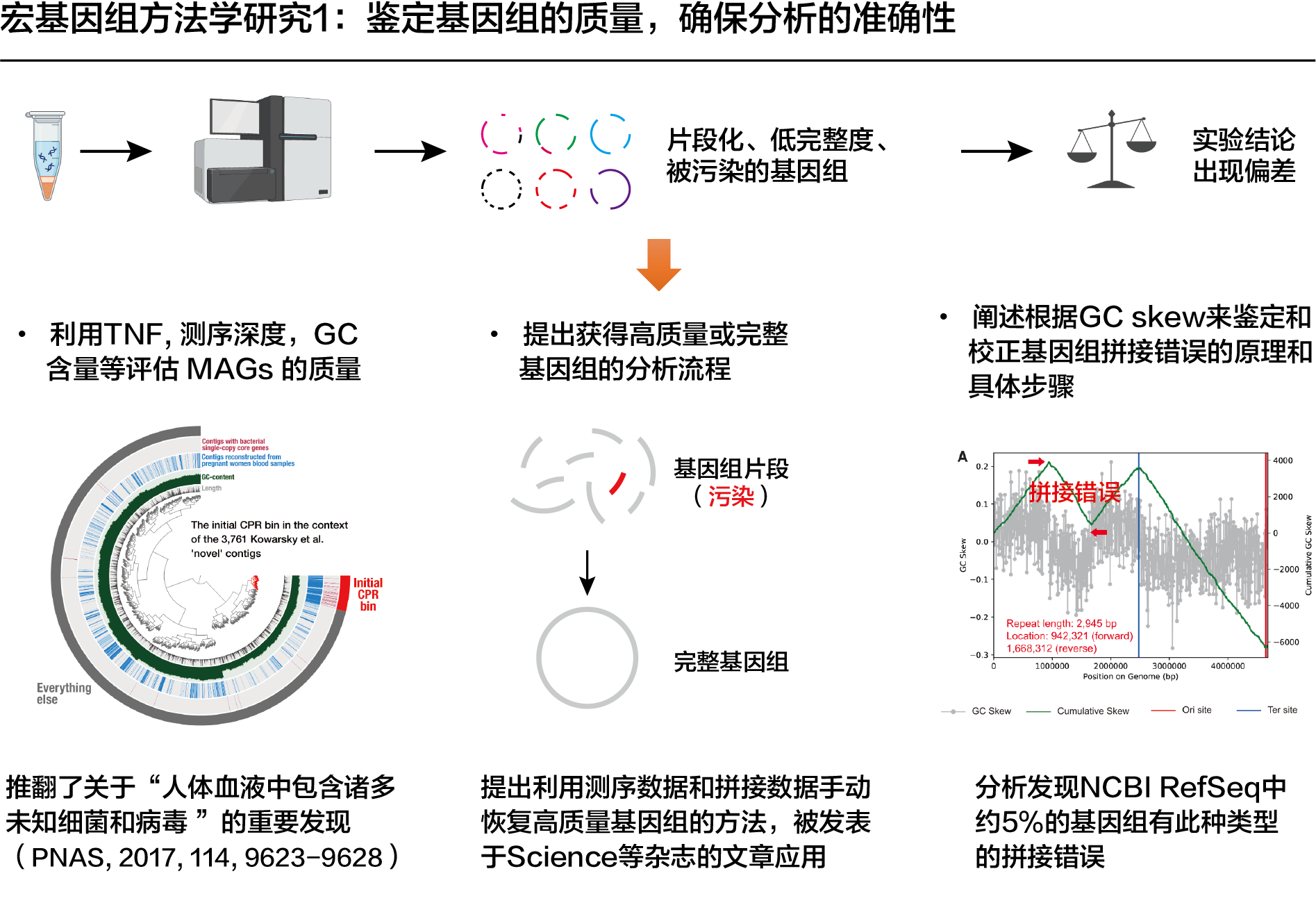
(1)鉴定基因组的质量,确保分析的准确性
针对宏基因组获得的 MAGs 通常是片段化、不完整和被污染的,提出了利用宏基因组片段的四核苷酸频率、测序深度和 GC 含量等特征来进行基因组分箱和评估 MAGs 质量的重要性。基于该方法,推翻了美国工程院院士 Stephen R. Quake 关于“人体血液中包含诸多未知细菌和病毒 ”的研究发现(PNAS 2017, 114, 9623–9628),因其未能正确地进行基因组分箱而高估了微生物的多样性。同时,建立了从宏基因组学数据中获得高质量基因组的方法流程,可将一般质量的基因组优化成高质量基因组;或将高质量但不是完整的基因组进行优化,以获得环状的没有任何拼接错误的基因组,因此可以完全确定微生物和病毒的基因组大小和基因顺序等特征。此外,基于基因组的 GC skew 图谱,发现如 NCBI RefSeq 等公共数据库中的完整微生物基因组(约5%)存在拼接错误,并建立了检测和纠正这些错误的详细方法流程。考虑到 RefSeq 中的基因组通常被作为“金标准”用于日常的分析和研究,该方法的建立为同行科学研究和分析的可靠性做出了重要贡献。这一工作以第一作者身份发表于Genome Research(2020)。
(2) 开发病毒基因组优化工具
病毒基因组的研究通常都基于单个的宏基因组拼接片段,而它们往往只能代表部分的基因组,因此对病毒的多样性、代谢潜能、进化历史、基因组结构和大小等的评估都存在偏差。近几年来,学界开发了一些针对病毒的基因组分箱工具,如 vRhyme 和 CoCoNet等,然而获得的病毒 MAGs 的质量却很难被准确评估(如 CheckV 等)。因此,根据常用的第二代测序数据拼接软件大都基于 de Bruijn graph 这一特点,开发了 COBRA 。COBRA 通过分析基因组片段的测序深度和双端 reads 的映射关系,来评估它们之间的位置关系,并对来自同一种群的片段进行连接,以获得高质量或完整的基因组(图5)。
Benchmarking 分析显示 COBRA 可将多达数十条基因组片段进行连接,得到一个完整的环状基因组。与常用的基因组分箱工具进行比较分析,结果显示 COBRA 能够以更高的准确度获得更多的高质量基因组;而分箱工具得到的40-80%病毒 MAGs 都有不同程度的污染,因此会直接影响下游分析的准确性。然后,利用 COBRA 对231个淡水宏基因组进行了分析,获得了7334个种水平的噬菌体基因组,其中超过6000个是从未被报道的。此分析同时报道了267个大噬菌体基因组,包括目前已知的第二大噬菌体基因组(717kbp)。对大噬菌体进行比较基因组分析表明,结构蛋白和 DNA 代谢相关的基因保持了高度的核苷酸相似性,而其他基因的获得或丢失是推动它们分化的主要因素。这些结果扩展了学界对淡水环境噬菌体和大噬菌体的多样性和进化历史的认识。通过对这些噬菌体的辅助代谢基因分析,发现约3.1%的基因组编码了与同化硫酸盐还原过程相关的基因(cysH 或 cysC),这表明淡水环境中的噬菌体可能在硫循环的过程具有重要作用。宏转录组数据的分析支持了这一推论,在某些富氧环境样品中,噬菌体编码的 cysC 基因的总转录活性高于微生物编码的该基因的活性。上述分析表明 COBRA 具有强大的基因组优化能力,能通过提供更多高质量的病毒基因组,从而确保分析的准确性,促进关于病毒的科学研究。上述工具以第一作者和共同通讯作者发表于Nature Microbiology(2024)。